Biologia & Ciências
13/08/2015
Bioinformática e Genoma
Núcleo usa bioinformática para investigar problemas das ciências genômicasPor Aline Naoe (USP Online)
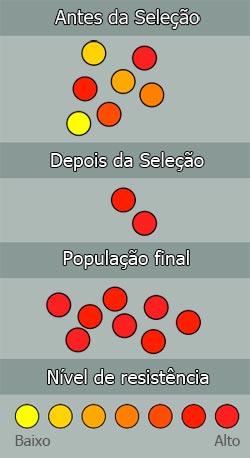
Exemplo de seleção natural: desenvolvimento de resistência a antibióticos em micro-organismos
Objetos de estudo tão diversos como uma bactéria que causa doenças em laranjeiras, câncer de próstata e diversidade microbiana em um zoológico estão reunidos em um mesmo núcleo de pesquisa na USP. O que aproxima estes projetos é a necessidade de processamento de numerosos dados de genômica, possível somente com o uso de recursos sofisticados da computação.
O Núcleo de Pesquisa em Biologia Computacional e Genômica (Nubic), coordenado pelo professor do Instituto de Química (IQ) da USP João Carlos Setubal, surgiu com o intuito de integrar os pesquisadores que desenvolvem ferramentas de informática voltadas a questões biológicas e os cientistas que precisam delas. “Desta forma as áreas podem conversar e, em alguns casos, ver que têm problemas em comum. Faz sentido ter um bioinformata trabalhando tanto com a expressão gênica em câncer de próstata como com doença em pé de laranja porque o dado é da mesma natureza e o processamento é muito parecido, às vezes até mesmo igual”, afirma.
A organização do Nubic, que é um dos Núcleos de Apoio à Pesquisa (NAPs) da USP, apoiados pela Pró-Reitoria de Pesquisa, se dá por meio dos chamados “projetos motores”. O coordenador explica que a articulação foi pensada desta forma porque, para ele, é preciso primeiro um projeto biológico para então motivar o desenvolvimento das ferramentas adequadas a ele. O grupo utiliza programas desenvolvidos em diferentes laboratórios de todo o mundo e, para os aspectos específicos de cada análise, cria também adaptações e novos programas.
Atualmente são sete os projetos motores, mas Setubal ressalta que esta configuração não é engessada, por estar em uma área muito dinâmica. “O Nubic é muito mais do que estes projetos. Cada integrante desenvolve várias atividades, que também estão no âmbito do Núcleo, apoiadas por ele, mesmo não constando na proposta original”. Com a criação do NAP, os pesquisadores adquiriram uma máquina sequenciadora de DNA e dois computadores de grande porte, o que representou um grande avanço em termos de infraestrutura que beneficiou todos os projetos.
Genética evolutiva
Um destes projetos envolve a análise da variação genética em populações humanas de todo o mundo, como africanos, europeus, asiáticos e nativos americanos. Liderado pelo professor Diogo Meyer, do Instituto de Biociências (IB) da USP, o estudo busca estabelecer comparações entre estas populações e enxergar a influência da seleção natural em sua genética. “Por exemplo, se uma mutação é muito comum na África, e muito rara no resto do mundo, há uma boa chance de que ela seja comum na África porque ela produz um efeito que favorece os indivíduos que vivem por lá”, explica.
Um estudo como esse, afirma Meyer, pode envolver a comparação de frequências de mutações em até 20 milhões de posições no genoma. “Para realizar tais tarefas precisamos de bioinformática. Isso sem falar nos testes estatísticos que são usados para responder à questão sobre seleção, que frequentemente incluem ferramentas estatísticas que demandam muitos recursos informáticos”.
Atualmente, o projeto envolve também o estudo de uma população específica, os quilombolas do Vale do Ribeira, região localizada entre o sul do estado de São Paulo e o norte do estado do Paraná. O intuito é verificar que tipo de mistura genética originou a população e como esta mistura é influenciada pela seleção natural.
Microbiomas do zoológico
O coordenador do Nubic lidera um projeto motor que analisa as bactérias presentes no processo de compostagem do Zoológico de São Paulo. Segundo Setubal, muitos detritos orgânicos são gerados no local, seja pelos animais ou pela própria mata. Por isso, o zoológico implantou um sistema de compostagem em larga escala. A degradação ocorre graças a certas espécies de bactérias presentes no ambiente que são capazes de digerir diferentes tipos de material orgânico, transformando-os em adubo que será usado pelo próprio zoológico.
O projeto trabalha com uma técnica chamada metagenômica, que permite analisar os DNAs das bactérias sem precisar isolá-las. Desta forma, em vez de selecionar apenas um tipo de bactéria presente em uma determinada amostra para então analisá-la, o pesquisador extrai o DNA diretamente da amostra. Com essa técnica, os DNAs de todas as espécies presentes na amostra vêm misturados, e a bioinformática é necessária para diferenciá-los. Esse tipo de estudo permite ter uma visão mais ampla do processo de degradação e entender quais bactérias agem, e de que forma, em cada etapa deste processo. “É uma pesquisa que tem o potencial de produzir descobertas importantes de natureza biotecnológica. A degradação de biomassa é entendida como o grande futuro para a geração de biocombustíveis”, afirma.
A lista completa dos projetos motores do Nubic e dos participantes do núcleo está disponível no site. Além de professores do IQ e do IB, o grupo reúne pesquisadores de outras unidades da USP, como o Instituto de Matemática e Estatística (IME), a Faculdade de Medicina de Ribeirão Preto (FMRP) e a Escola de Artes, Ciências e Humanidades (EACH), além de outras instituições nacionais e internacionais.
Esta notícia foi publicada na Edição Online de 05 de agosto 2015 da revista USP Online Destaque. Todas as informações nela contida são de responsabilidade do autor.

